Como si se tratara de hackers tomando el control de una red de ordenadores, algunas bacterias nos infectan inyectando sus propios elementos perniciosos en nuestras células. Estas moléculas “maliciosas”, llamadas efectores, toman el control de nuestras células, bloqueando las señales de alarma necesarias para avisar al sistema inmune. Esto permite a las intrusas colonizar nuestro cuerpo. Desgraciadamente, los antibióticos son poco eficaces contra estas enfermedades. Un reto abierto es encontrar tratamientos alternativos y, para ello, es clave comprender cómo funciona este proceso infectivo. Pero ésta no es una tarea sencilla. Las bacterias cuentan con una gran variedad de efectores y su interacción con los componentes de las células es realmente compleja.
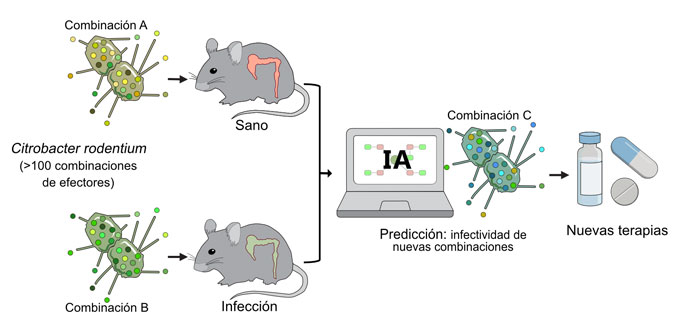
A causa de esta enorme complejidad, hasta ahora no se había estudiado cómo estas moléculas, los efectores bacterianos, trabajan conjuntamente dentro de nuestras células. Ahora, un equipo internacional de científicos españoles, del Reino Unido y de Israel, en el que participa la Universidad Politécnica de Madrid (UPM), han aunado fuerzas para llevar a cabo la titánica labor de analizar todas estas moléculas de forma conjunta. Para conseguirlo, ha sido necesario combinar experimentos de laboratorio y herramientas de Inteligencia Artificial (IA). En este trabajo pionero, publicado en la revista Science, han utilizado ratones infectados con 100 variantes de la bacteria Citrobacter rodentium, cada una con un repertorio distinto de efectores.
Los investigadores de la UPM, el catedrático de IA, Alfonso Rodríguez-Patón y la estudiante de doctorado, Elena Núñez Berrueco, utilizaron los datos recopilados en el laboratorio para construir un modelo de aprendizaje automático, usando novedosas técnicas de IA. El número de combinaciones posibles de efectores supera los mil millones, por lo que estudiar todas las variantes supondría más de mil años de investigación experimental. Ahí es donde entra en juego la IA para cambiar las reglas y permitir descifrar este complejo mecanismo. El algoritmo desarrollado en la UPM es capaz de predecir la capacidad infectiva de cualquier variante tras aprender los patrones de los 100 experimentos de laboratorio.
Elena Núñez Berrueco afirma que “al estudiar un sistema biológico tan complejo, la IA es capaz de ver lo que no es evidente ante nuestros ojos. Las predicciones nos ayudan a identificar las combinaciones de efectores más relevantes y así ahorrar tiempo y recursos. Podemos usar este modelo para predecir si una nueva cepa, con una combinación de efectores diferente a las estudiadas, puede manipular a nuestras células y la forma en que lo hace.”
El algoritmo desarrollado está inspirado en las redes de neuronas artificiales, pero incorpora conocimiento extraído de la literatura sobre las dianas objetivo de los efectores. La arquitectura de esta red tiene una particularidad: en lugar de ser genérica, posee la misma forma que la red de interacciones biológicas de los efectores con los componentes de nuestras células. Esto ha permitido entrenar la red con un número muy reducido de casos, dando lugar, además, a un modelo con resultados interpretables (la tan deseada “IA explicable”).
Con la ayuda del modelo, los científicos han podido dirigir los siguientes experimentos hacia las variantes más interesantes. Así, han podido descubrir pequeños grupos de estas moléculas que son esenciales. Esto significa que, cuando se eliminan o bloquean, las bacterias no infectan, suponiendo una prometedora diana para futuros tratamientos que nos ayuden a vencer a estos hábiles invasores.
El catedrático Alfonso Rodríguez-Patón continúa: «La Inteligencia Artificial se muestra una vez más como una tecnología disruptiva. En este caso, en el campo de la microbiología. Esta investigación interdisciplinar, nos ha exigido desarrollar novedosas técnicas de IA para desentrañar la compleja red de señales moleculares que las bacterias emplean para infectarnos. Los resultados obtenidos son muy satisfactorios por lo que continuaremos colaborando con el grupo de Gad Frankel en el Imperial College de Londres en futuras investigaciones.»
Ref: David Ruano-Gallego, Julia Sánchez-Garrido, Zuzanna Kozik, Elena Núñez-Berrueco, Massiel Cepeda-Molero, Caroline Mullineaux-Sanders, Jasmine Naemi-Baghshomali Clark, Sabrina Slater, Naama Wagner, Izabela Glegola-Madejska, Theodoros I. Roumeliotis, Tal Pupko, Luis Ángel Fernández, Alfonso Rodríguez-Patón, Jyoti S. Choudhary and Gad Frankel. “Deconstructing a type III secretion system effector network unravels the inherent robustness and plasticity in pathogenesis and immunity” https://doi.org/10.1126/science.abc9531
Fuente: UPM





